
-

生物通官微
陪你抓住生命科技
跳动的脉搏
Nature子刊丨从宏基因组文库中高效检索目标序列的新方法
【字体: 大 中 小 】 时间:2023年02月09日 来源:
编辑推荐:
该方法将PacBio HiFi测序与CRISPRi技术结合,可从复杂文库中高效检索目标克隆,加速基因组信息的挖掘,为研究宏基因组微生物多样性提供了更多的可能性,并有望扩展到合成生物学等重要领域。

生命科学领域中一种新方法的创建往往需要至少一种成熟技术的支撑,高通量PacBio HiFi测序技术凭借其长读长且准确性高的特点与多种技术手段结合,获得高效准确的方法被应用到微生物、动植物、医学以及合成生物学领域中,获得更完整视野分析。
宏基因组是环境微生物群落中所有微生物物种基因组的集合,且包含数千万个独特的克隆,数量庞大,但绝大部分微生物的不可培养性使微生物多样性的研究以及开发利用受到了限制。一直以来,从大型宏基因组文库中靶向检索克隆使用的是基于多步稀释和聚合酶链式反应(PCR)的筛选方法,此方法费时费力。虽然CRISPR/Cas技术已被用于检索克隆目标基因序列,但还未被验证能从复杂的宏基因组库中检索序列。
知名期刊Nature Biotechnology 近期发表了题为High-throughput retrieval of target sequences from complex clone libraries using CRISPRi的文章,该文章提出了一种适用于研究宏基因组微生物多样性的新方法——利用核酸酶缺陷型 Cas9 (dCas9)构建的反选择中断回路( CRISPR counter-selection interruption circuit,CCIC),该方法将PacBio HiFi测序与CRISPRi技术结合,可从复杂文库中高效检索目标克隆,加速基因组信息的挖掘,为研究宏基因组微生物多样性提供了更多的可能性,并有望扩展到合成生物学等重要领域。

什么是CCIC方法?
CICC法是以CRISPR干扰(CRISPRi)技术为基础,利用dCas9可进行序列特异性定位的功能,通过阻断启动子或转录延伸来抑制基因表达。由于sacB基因编码蔗糖果聚糖酶,该酶能催化蔗糖水解成葡萄糖和果糖,并且将果糖聚合成高分子量的果聚糖,但高分子量果聚糖积累对细胞存在潜在的毒性作用,可造成细胞死亡,所以利用蔗糖致死基因sacB在菌株中是否正常表达作为反向选择的依据(图1)。以P1派生人工染色体(P1-derived artificial chromosome,PAC)/粘粒作为载体,将sacB基因与其强组成型启动子以及两者之间的多克隆位点、插入的DNA片段共同组装到PAC载体中。其中在多克隆位点处插入了预先设计的sgRNA可以靶向结合的简并序列(条形码)。借助于Pacbio HiFi长读长测序技术,在CICC文库中获得准确的条形码与插入的DNA片段的对应关系。当需要在CICC文库中检索序列时,利用dCas9靶向结合条形码序列阻碍sacB的表达,从而获得在蔗糖环境可以存活的菌株,即为包含目标序列的菌株(图2)。进一步测试该方法筛选目标序列的效率,作者构建了三个分别含有5,000, 50,000 和 100,000个非目标PACs文库,从中筛选一种目标PAC,结果表明,含有50,000个非目标序列的混合文库中目标序列的阳性命中率高于70%。
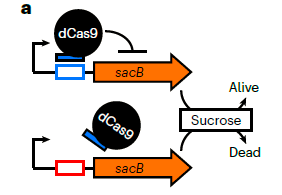
图1. dCas9 与 sgRNA (蓝色填充的黑色方框) 靶向结合sacB基因启动子下游序列 (无颜色填充的蓝色方框),抑制sacB表达,在蔗糖培养基上的细胞存活;如dCas9 无法结合sacB基因启动子下游序列(无颜色填充的红色方框),在蔗糖培养基上的细胞死亡。
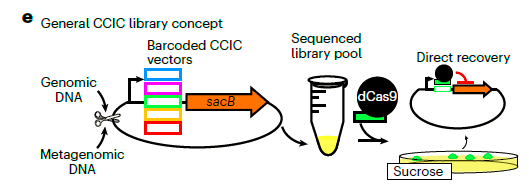
图2. 基因组/宏基因组DNA克隆到含有条形码、sacB以及启动子的载体中构建CCIC文库,通过测序获得文库中DNA插入片段和条形码的对应关系,最后通过dCas9介导抑制sacB基因的表达,获得蔗糖培养基中存活的菌株。
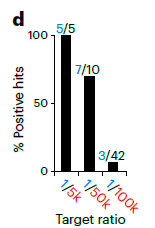
图3. 含有靶向序列的PAC与不含靶向序列的PAC以1/5k、1/50k、1/100K不同比例混合。
被鉴定出的目标序列克隆以蓝色数字表示,不含靶向序列的PAC克隆以黑色数字表示。
CCIC方法应用于宏基因文库中进行DNA检索
为了满足大片段宏基因DNA大型克隆文库的筛选,以λ噬菌体包裹粘粒作为载体应用于宏基因组文库中序列的检索。作者从土壤中提取基因组DNA并构建10000个粘粒文库(支持插入31 kb—46 kb的DNA片段)。使用PacBio HiFI测序技术对构建好的粘粒文库进行分析获得条形码序列和插入的DNA片段的对应关系,通过antiSMASH 和DefenseFinder两种预测工具分析进一步获得感兴趣的生物基因合成簇(biosynthetic gene clusters,BGCs)和噬菌体防御系统相关基因。通过PacBio HiFi测序技术获得4.73 Gbp数据,平均长度为7 kb,结合分析工具成功的分析出包含12类BGCs的66个粘粒文库以及包含4类CRISPR/Cas系统相关基因的4个粘粒文库。最后设计上述70个粘粒文库的sgRNA序列,利用dCas9介导进行反向筛选(图4)。目标检索效率可达到95%,同时CICC方法筛选目标克隆仅需2天时间,比以往的方法更加高效。同时CICC法支持31kb—46kb大片段DNA序列的筛选,结合长读长HiFi测序实属于强强联手。
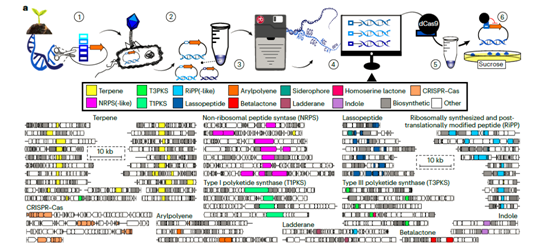
图4. 使用CCIC方法检索宏基因组文库中目标序列的基本流程。(1)将从土壤中获得的DNA克隆到含有条形码序列以λ噬菌体包裹的粘粒载体中(条形码序列以不同蓝色梯度的方框表示,sacB以橘色箭头表示)(2)在大肠杆菌中培养粘粒文库(3)使用PacBio HiFi CSS模式对粘粒文库进行测序(4)对测序获得的序列进行生物信息学分析鉴定文库的多样性以及与条形码序列的关系(5)将与条形码匹配的是sgRNA转入到文库中(6)触发dCas9介导sacB基因沉默,通过蔗糖培养基中特异性克隆的存活实现目标序列的检索。底部为使用CCIC方法检索到的4类CRISPR/Cas系统和12类生物基因合成簇。
无论是研究宏基因组文库微生物多样性还是在合成生物学研究中获得完整的BGCs信息,都需要对目标样本进行测序获得完整且准确的基因序列信息,“长且准”的PacBio HiFi测序不止一次的被应用到上述领域。
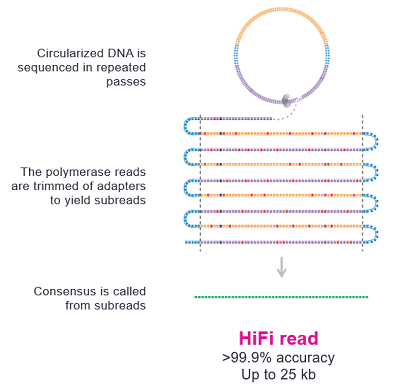
HiFi 测序是PacBio测序平台推出的兼顾长读长和高准确度的测序技术。作为PacBio最新的数据类型,既兼顾读长(10-25kb)又具有高准确度(单分子准确率>99.9%)的HiFi reads,不仅在合成生物学中基因组的挖掘应用中有着不错的表现,还可以实现全长16s rRNA测序,以更高的分辨率定义微生物种级别分类,构建完整的宏基因组组装基因组(MAGs)实现微生物与环境关系的分析,同时可以更准确的定义出功能基因,以便进一步的分析。
参考文献:High-throughput retrieval of target sequences from complex clone libraries using CRISPRi[J]. Nature Biotechnology.
DOI:https://doi.org/10.1038/s41587-022-01531-8
欲了解PacBio HiFi测序更多资讯,可添加下方企业微信或联系您身边的基因人。

今年成立三十周年的基因有限公司作为PacBio公司在中国区的独家代理商,自2011年以来将PacBio第三代单分子实时测序技术引入国内,一直为国内用户提供专业的三代测序系统的安装培训,技术支持,应用培训与售后维护工作,赢得客户的一致好评与信任。基因有限公司将一如既往的支持越来越多的PacBio用户。

下载安捷伦电子书《通过细胞代谢揭示新的药物靶点》探索如何通过代谢分析促进您的药物发现研究
10x Genomics新品Visium HD 开启单细胞分辨率的全转录组空间分析!
 生物通微信公众号
生物通微信公众号
知名企业招聘
今日动态 | 人才市场 | 新技术专栏 | 中国科学人 | 云展台 | BioHot | 云讲堂直播 | 会展中心 | 特价专栏 | 技术快讯 | 免费试用
版权所有 生物通
Copyright© eBiotrade.com, All Rights Reserved
联系信箱:
粤ICP备09063491号
