
-

生物通官微
陪你抓住生命科技
跳动的脉搏
邢栋课题组利用单细胞多组学技术揭示三维基因组与基因表达的关系
【字体: 大 中 小 】 时间:2023年06月20日 来源:北京大学新闻网
编辑推荐:
2023年6月9日,北京大学生物医学前沿创新中心(BIOPIC)邢栋课题组在Science期刊发表了题为“Linking genome structures to functions by simultaneous single-cell Hi-C and RNA-seq”的研究论文,报道了一种新型单细胞多组学技术HiRES(Hi-C and RNA-seq em...

在高等真核生物中,基因组在细胞核内的三维空间构象对细胞功能至关重要。例如,增强子常常通过三维空间相互作用对远端目的基因的转录活性起调控作用1-3。三维基因组水平上的异常也被发现与包括癌症在内的多种疾病的发生密切相关4-6。然而,三维基因组与基因表达的整体关系仍然存在争议。例如,通过靶向降解染色质空间构象的关键调控蛋白CTCF或cohesin,可以使基因组空间构象发生重大重排,但对基因表达却只造成了较微弱的影响7-9。在果蝇胚胎中,不同细胞类型间的基因表达具有显著差异,但染色质结构差别并不明显10,11。因此,想要进一步认识复杂组织器官和丰富细胞类型背景下的染色质三维结构和基因表达之间的关系,亟需更有力的工具。
2023年6月9日,北京大学生物医学前沿创新中心(BIOPIC)邢栋课题组在Science期刊发表了题为“Linking genome structures to functions by simultaneous single-cell Hi-C and RNA-seq”的研究论文,报道了一种新型单细胞多组学技术HiRES(Hi-C and RNA-seq employed simultaneously),首次基于测序方法实现了在单细胞水平对转录组和三维基因组的同时检测。
HiRES方法在细胞群体水平进行原位反转录和染色质构象捕获(3C),之后通过流式分选得到单细胞,再对每个单细胞进行扩增后测序(图1A)。DNA或RNA读段通过反转录过程中引入的RNA识别序列进行区分,因此该方法不涉及DNA和RNA的物理分离,最大程度地提高了检测效率。在小鼠大脑样品中,平均每个细胞可以检测到6517个基因,27,468条转录本(UMIs)以及317,435个染色质相互作用(图1B、C)。利用该数据,研究人员可以对单细胞三维基因组进行高分辨率结构重构,并可以在三维结构上对基因表达水平进行观测(图1D、E)。
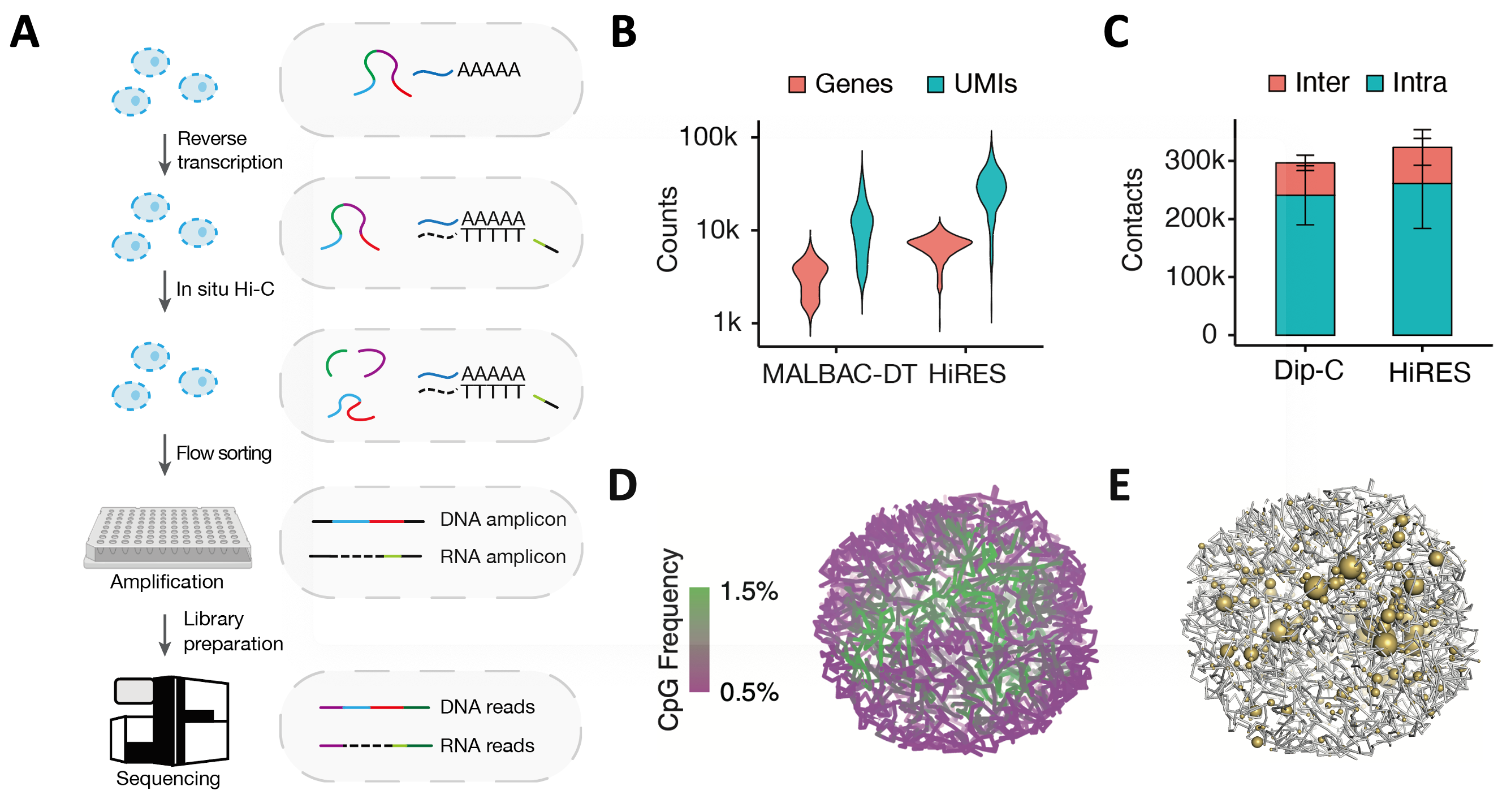
图1 HiRES方法能高效检测单细胞的转录组和三维基因组(A)HiRES方法流程示意图。(B)MALBAC-DT和HiRES两种方法检测到的基因和转录本数目。(C)Dip-C和HiRES两种方法检测到的染色质相互作用数。(D)示例单细胞染色质三维结构上的染色质区室化情况,可以看到异染色质(洋红色)和常染色质(绿色)的分区。(E)示例单细胞染色质三维结构上的基因表达情况。球的大小代表表达量的高低
之后,利用HiRES方法,研究人员绘制了小鼠胚胎自妊娠后7天至11.5天这一时期共7469个单细胞的转录组和三维基因组图谱。通过对该双组学数据的分析,该研究在以下几个方面进行了探索:
一、不同细胞类型中细胞周期对三维基因组的影响
胚胎发育时期的细胞处在快速增殖的过程中,细胞周期对三维基因组的影响不容忽视。该研究利用HiRES的双模态数据,开发了一种对单细胞进行细胞周期划分的策略。该策略主要依赖DNA空间相互作用特征、细胞周期相关基因表达以及DNA复制程度等指标,可以将单细胞分配到不同的细胞周期状态之一(图2)。
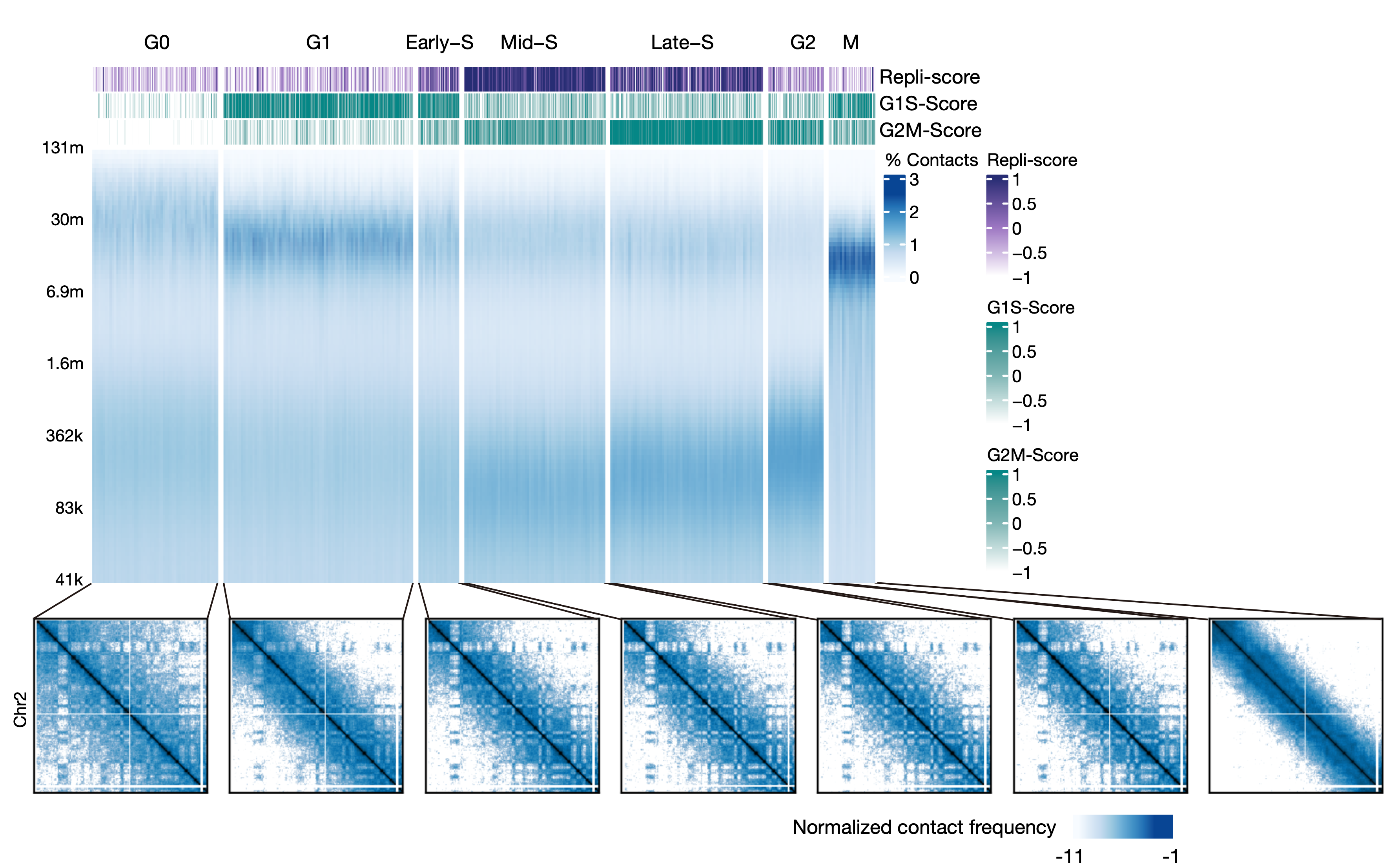
图2 利用双组学特征注释细胞周期状态。图中每一列为一个单细胞
在胚胎组织中,研究人员发现根据染色质结构定义的细胞类群与细胞周期状态之间的一致性更强,说明与细胞周期相关的染色质结构变化对总体的染色质空间构象具有重要影响。在不同的细胞类型之间,该研究开发了一种半定量的比较细胞周期过程中染色质结构的方法,并发现分裂间期细胞的染色质构象由两个独立的过程决定——有丝分裂后染色体的自发展开和基因组的复制。对于G1期较短的细胞类型,这两个过程在时间上可能重叠,从而共同决定了染色质三维结构的细胞周期动态。
二、不同细胞类型间的差异染色质相互作用与细胞功能密切相关
研究不同细胞类型的染色质结构差异是三维基因组研究的重要问题之一。利用Hi-C技术寻找差异染色质相互作用常常面临数据的稀疏性、高噪音和较少的生物学重复等困难。针对这些问题,该研究设计了Simple Diff策略,通过对两组重构的单细胞全基因组三维结构中两点之间的空间距离进行非参检验的方式,可以高效检测染色质差异相互作用(differential interaction,DI)。基于该方法,研究人员发现染色质结构的特化在原肠形成时期就已经出现,并且差异染色质结构位点上显著富集了差异表达基因,提示染色质结构变化与细胞功能紧密相关。
在此基础上,该研究进一步利用染色质相互作用强度与基因表达水平的协同变化,将差异相互作用与其潜在的相关基因联系起来,并将这些与基因表达显著相关的差异相互作用称为GADI(gene-associated DI)。研究发现GADI常常将启动子与远端的调控元件(如增强子、超级增强子)连接起来(图3)。因此,GADI不仅仅是转录活动的结果,还包含了许多潜在的增强子-启动子相互作用,这些相互作用很可能是控制细胞类型特异性基因表达的基础。
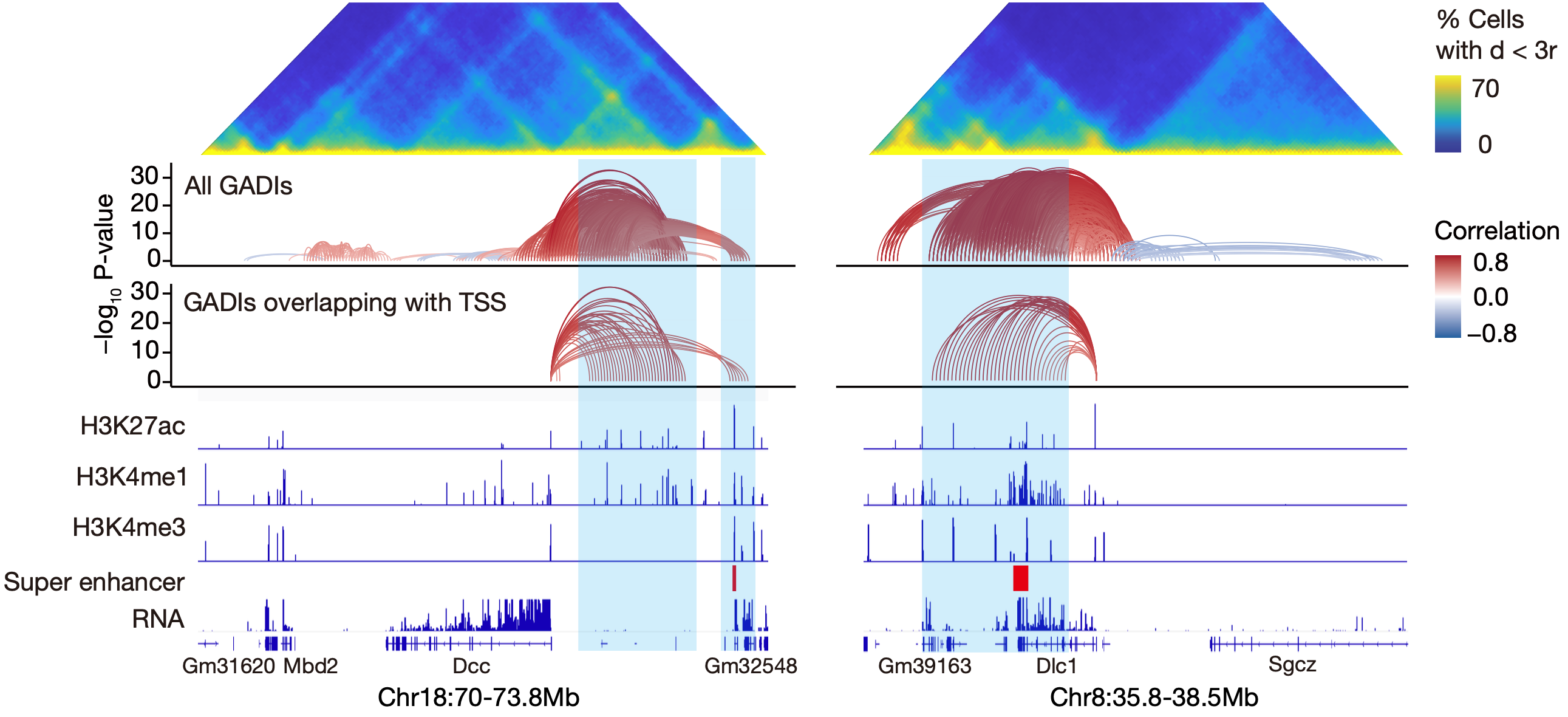
图3 连接到Dcc和Dlc1基因的GADI示例
三、发现广泛存在的先于基因转录激活的染色质结构重排
在将染色质相互作用与特定基因联系起来后,该研究进一步比较了染色质构象变化和转录水平变化在拟时间序列上的先后关系,并鉴定了超过4万个先于转录变化的染色质相互作用变化。通过对这些染色质空间构象变化位点的表观遗传学分析,发现这种先于转录的染色质构象变化主要分为两类:首先,在大多数基因位点上,先于基因表达的染色质构象重排主要建立在活性染色质之间,这可能是增强子的激活所驱动的;其次,对于一部分位于抑制性染色质环境中的基因,转录激活前需要出现异染色质结构的松弛(图4)。尽管这两种过程背后更详细的分子机制有待进一步探索,这一结果强烈地支持了染色质三维结构广泛参与基因表达调控的观点。
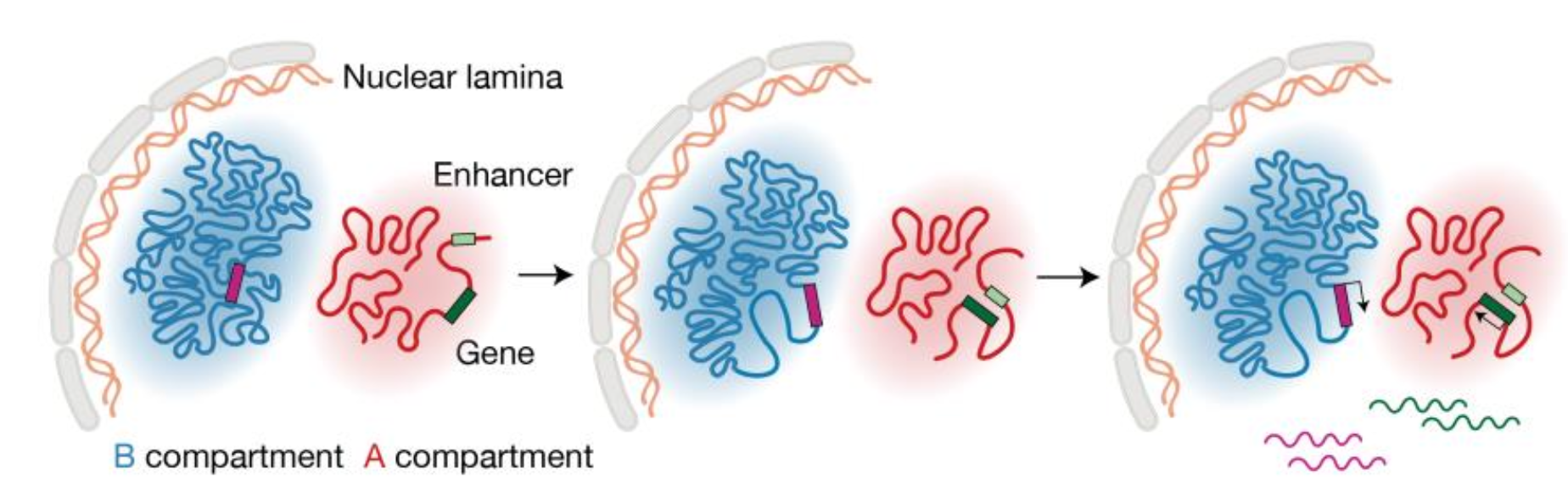
图4 转录激活前发生的染色质结构重构的示意图
北京大学生物医学前沿创新中心、生命科学学院博士生刘致远、陈玉洁、夏启旻为该论文的共同第一作者,邢栋为该论文通讯作者。该研究得到了科技部、国家自然科学基金、北京大学"临床医学+X-青年学者项目"和北京未来基因诊断高精尖创新中心的资助,以及北京大学高通量测序平台的协助与支持。
参考文献:
1 Bas Tolhuis, R.-J. P., Erik Splinter, & Frank Grosveld, a. W. d. L. Looping and Interaction between Hypersensitive Sites in the Active beta-globin Locus. Molecular Cell (2002).
2 Deng, W. et al. Reactivation of developmentally silenced globin genes by forced chromatin looping. Cell158, 849-860 (2014). https://doi.org:10.1016/j.cell.2014.05.050
3 Tang, Z. et al. CTCF-Mediated Human 3D Genome Architecture Reveals Chromatin Topology for Transcription. Cell163, 1611-1627 (2015). https://doi.org:10.1016/j.cell.2015.11.024
4 Lupianez, D. G. et al. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell161, 1012-1025 (2015). https://doi.org:10.1016/j.cell.2015.04.004
5 Flavahan, W. A. et al. Insulator dysfunction and oncogene activation in IDH mutant gliomas. Nature529, 110-114 (2016). https://doi.org:10.1038/nature16490
6 Xu, Z. et al. Structural variants drive context-dependent oncogene activation in cancer. Nature612, 564-572 (2022). https://doi.org:10.1038/s41586-022-05504-4
7 Nora, E. P. et al. Targeted Degradation of CTCF Decouples Local Insulation of Chromosome Domains from Genomic Compartmentalization. Cell169, 930-944 e922 (2017). https://doi.org:10.1016/j.cell.2017.05.004
8 Rao, S. S. P. et al. Cohesin Loss Eliminates All Loop Domains. Cell171, 305-320 e324 (2017). https://doi.org:10.1016/j.cell.2017.09.026
9 Wutz, G. et al. Topologically associating domains and chromatin loops depend on cohesin and are regulated by CTCF, WAPL, and PDS5 proteins. EMBO J36, 3573-3599 (2017). https://doi.org:10.15252/embj.201798004
10 Ing-Simmons, E. et al. Independence of chromatin conformation and gene regulation during Drosophila dorsoventral patterning. Nat Genet53, 487-499 (2021). https://doi.org:10.1038/s41588-021-00799-x
11 Espinola, S. M. et al. Cis-regulatory chromatin loops arise before TADs and gene activation, and are independent of cell fate during early Drosophila development. Nat Genet53, 477-486 (2021). https://doi.org:10.1038/s41588-021-00816-z
 生物通微信公众号
生物通微信公众号