
-

生物通官微
陪你抓住生命科技
跳动的脉搏
生命科学学院陈铭团队在Nucleic Acids Research发文提出一个具有潜在ncPEP信息和细胞类型特异性相互作用的植物ncRNA数据库
【字体: 大 中 小 】 时间:2024年11月05日 来源:浙江大学生命科学学院
编辑推荐:
近年来,植物非编码RNA(ncRNAs)领域取得了重大进展,许多ncRNAs被认为是植物发育和胁迫反应中基因表达的重要调节因子。此外,这些ncRNA的编码潜力,产生ncRNA编码肽(ncPEPs),已成为一个重要的研究领域。然而,现有的植物ncRNA数据库缺乏关于ncRNA编码肽(ncPEPs)和细胞类型特异性相互作用的全面信息。2024年10月29日,浙江大学陈铭团队在Nucleic Acids Research 在线发表题为“ncPlantDB: a plant ncRNA database with ...

近年来,植物非编码RNA(ncRNAs)领域取得了重大进展,许多ncRNAs被认为是植物发育和胁迫反应中基因表达的重要调节因子。此外,这些ncRNA的编码潜力,产生ncRNA编码肽(ncPEPs),已成为一个重要的研究领域。然而,现有的植物ncRNA数据库缺乏关于ncRNA编码肽(ncPEPs)和细胞类型特异性相互作用的全面信息。
2024年10月29日,浙江大学陈铭团队在Nucleic Acids Research 在线发表题为“ncPlantDB: a plant ncRNA database with potential ncPEP information and cell type-specific interaction”的研究论文,该研究提出了ncPlantDB,一个综合数据库,整合了43种植物的ncRNA和ncPEP数据。
ncPlantDB包含353 140个ncRNAs、3799个ncPEPs和4 647 071个相互作用,来源于已建立的数据库和文献挖掘。该数据库提供了独特的功能,包括翻译潜力数据、源自单细胞RNA测序和Ribo-seq分析的细胞特异性相互作用网络以及交互式可视化工具。ncPlantDB提供了一个用户友好的界面,用于在单细胞水平上探索ncRNA表达模式,促进组织特异性ncRNA和潜在ncPEP的发现。通过整合不同的数据类型并提供先进的分析工具,ncPlantDB为研究植物ncRNA功能、相互作用及其潜在编码能力的研究人员提供了宝贵的资源。该数据库显著增强对植物ncRNA生物学的理解,并为探索植物基因组学中复杂的调控网络开辟了新的途径。

非编码RNA(ncRNAs)在植物生物学中起着至关重要的调节作用,影响发育、胁迫反应和适应。最近的进展强调了ncRNAs通过复杂的分子机制介导这些过程的重要性。除了它们的调节功能外,许多ncRNAs还具有通过其小的开放阅读框(sORFs)编码小肽的能力。这些肽被称为ncRNA编码肽(ncPEPs),由5-60个氨基酸组成,提供了意想不到的基因调控和蛋白质功能层。自发现miRNA编码的调控肽miPEP171b和miPEP165a以来,这一概念最近已扩展到其他miRNA和其他ncRNA类型。miPEPs由pri-miRNA 5'区的第一个开放阅读框(miORF)编码,可增强其原始pri-miRRNA的转录。此外,这些NCPEP已被逐步证明具有可持续农业的潜力。因此,纳入ncPEPs提供了宝贵的见解,并扩展了我们对ncRNAs在植物生物学中的多方面作用的理解。
单细胞技术的出现极大地改变了我们对非编码 RNA(ncRNAs)的认知,使我们能够在单细胞水平上深入探究它们的表达和调控网络。这使得对细胞特异性 ncRNA 功能及其对细胞异质性的贡献能够进行细致剖析,而这对于理解植物复杂的生物过程至关重要。单细胞网络分析提升了我们描绘 ncRNAs 在不同细胞类型和发育阶段精确调控作用的能力,为基础植物生物学研究和农业应用提供了关键见解。现有的部分 ncRNA 数据库,存在对 ncRNAs 潜在编码能力关注不足的问题,也未纳入用于表达和相互作用分析的单细胞数据。
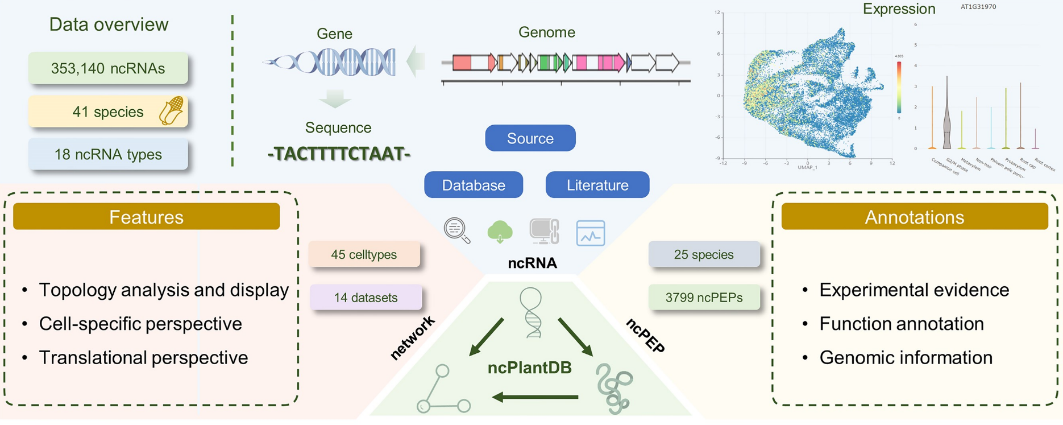
为了解决这些局限性,研究人员开发了ncPlantDB。它整合了全面的数据集,提供翻译潜力信息,并结合单细胞数据分析,以实现对细胞和分子水平上非编码 RNA(ncRNA)功能的全面理解和探索。ncPlantDB 是研究人员不可或缺的资源,有助于深入探索植物基因组学中非编码 RNA 和非编码肽(ncPEP)的表达、相互作用和功能。预计 ncPlantDB 将为该领域做出重大贡献,并期待其被科学界广泛采用。
浙江大学生命科学学院陈铭教授为本文通讯作者,硕士研究生刘丽亚、博士研究生刘恩言为本文的共同第一作者。本研究得国家自然科学基金、国家重点研发计划与浙江省自然科学基金等项目的资助与支持。
原文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkae1017/7848845#
 生物通微信公众号
生物通微信公众号
知名企业招聘