
-

生物通官微
陪你抓住生命科技
跳动的脉搏
藏羚首个染色体水平的高质量基因组
【字体: 大 中 小 】 时间:2024年11月14日 来源:中科院高原生物所
编辑推荐:
相关研究结果以?A high-quality chromosome-level reference genome assembly of Tibetan antelope (Pantholops hodgsonii) 为题,于11月12日在Nature旗下综合性科学期刊Scientific Data(中科院二区)在线发表
藏羚(Pantholops hodgsonii)是世居青藏高原的典型反刍动物,平均分布海拔3,250-5,500 m,属鲸偶蹄目(Cetartiodactyla),牛科(Bovidae),藏羚属(Pantholops),是藏羚属唯一物种。19世纪中期至20世纪初,猖獗的非法盗猎使藏羚的种群数量下降了约90%,2000年被IUCN红色濒危物种名录评估为濒危物种。经过30多年的保护,藏羚的种群数量恢复到了20多万只,IUCN对其评级也从濒危降为近危。藏羚不仅是世界上分布海拔最高的反刍动物之一,也是青藏高原唯一具有长距离迁徙行为的物种,是研究高海拔适应性机制和迁徙行为的良好模型。然而迄今为止,公开数据库中仍缺少藏羚高质量的染色体水平基因组,严重限制了基于遗传特征解析其物种适应、进化及种群生态相关工作的开展。
中国科学院西北高原生物研究所动物生态与资源保护研究团队联合青海大学,基于PacBio HiFi三代基因组测序、Hi-C测序和DNBSEQ-T7二代基因组survey测序三种测序技术,成功组装了藏羚染色体级别的基因组。结果显示,藏羚基因组大小为3.1 Gb,Contig N50为84.6 Mb,所有基因挂载到30条染色体上(29 + X),与之前的核型研究一致。数据评估显示,藏羚基因组BUSCO得分为98.2%(S:92.3%,D:5.9%,F:0.8%,M:1.0%),平均QV值为70.14,表明组装的连续性好,完整度和准确性高。基于EDTA和RepeatModeler从头预测的藏羚基因组中重复序列注释结果表明,藏羚基因组重复序列主要由SINEs、LINEs、LTRs 和DNA transposons四种类型组成,序列总长度为1.65 Gb,占基因组的52.47%;基于蛋白同源预测、蛋白从头预测和深度学习等多种策略,在藏羚基因组上共注释到28,330个功能基因。综上,通过多种技术手段,首次获得了藏羚染色体水平的高质量基因组和注释信息,为藏羚的适应进化遗传机制、保护遗传学研究及进一步探索物种迁徙行为的遗传机制提供了重要的基因组资源。
相关研究结果以?A high-quality chromosome-level reference genome assembly of Tibetan antelope (Pantholops hodgsonii) 为题,于11月12日在Nature旗下综合性科学期刊Scientific Data(中科院二区)在线发表。西北高原所博士研究生徐波和青海大学陈家瑞副教授为共同第一作者,西北高原所张同作研究员和青海大学魏青副教授为共同通讯作者。该工作得到青海省自然科学基金团队项目(2023-ZJ-901T)的资助。
论文链接:https://www.nature.com/articles/s41597-024-04089-z
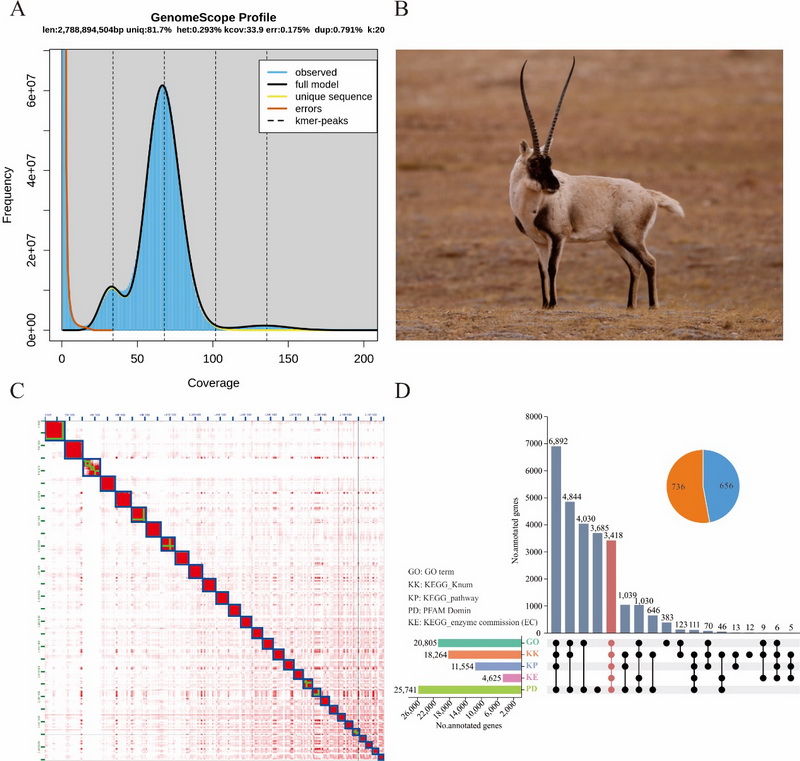
藏羚基因组的组装和注释结果
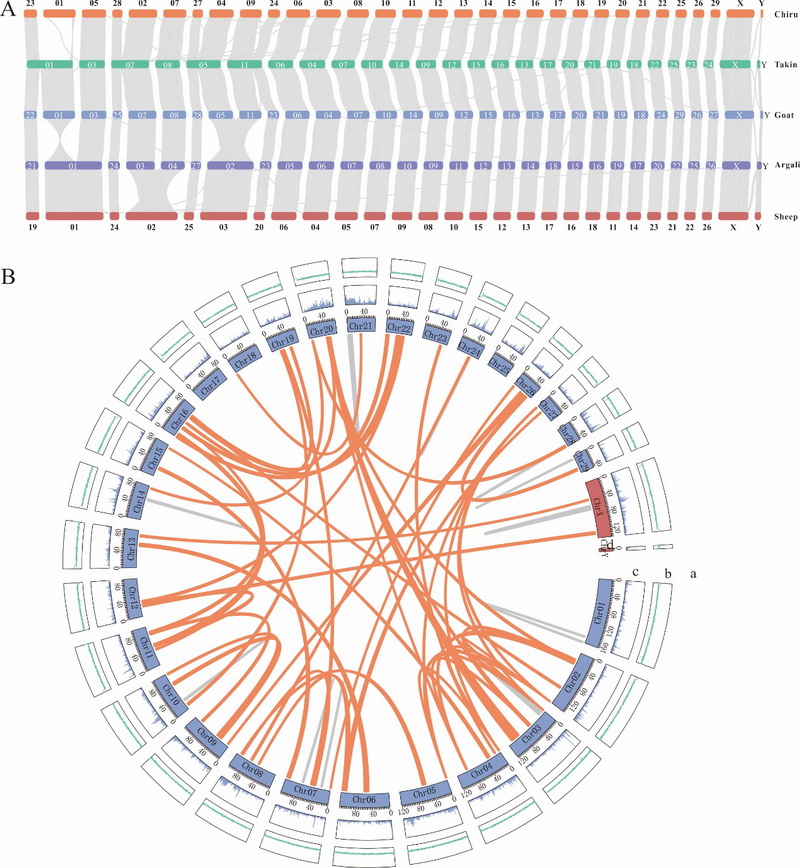
藏羚的基因组特征及与近缘物种之间的基因组共线性结果
 生物通微信公众号
生物通微信公众号
知名企业招聘